启动子
启动子是一段位于目标基因转录区上游、能够与RNA聚合酶结合从而实现转录起始的DNA序列[1]。在原核生物中,执行转录功能的转录机器—RNA 聚合酶全酶(包含α2、β、β'、ω、σ等亚基)依赖于其中的σ因子,特异性地识别具有特定序列特征的启动子区域并与之结合,从而引发转录的开始[2]。
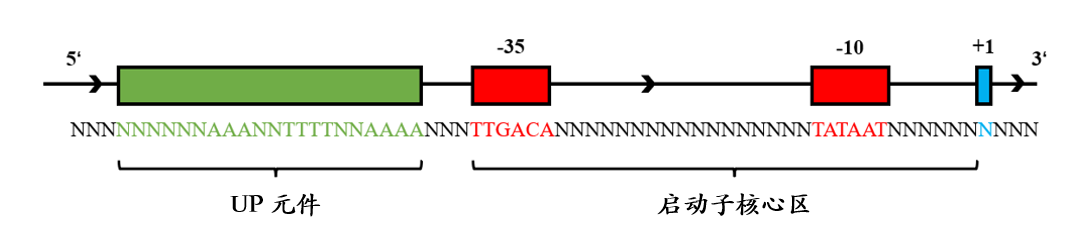
图1 原核微生物启动子序列特征示意图
枯草芽孢杆菌是一种安全性高的重要工业微生物,广泛应用于酶类、核苷、D-核糖、维生素、抗菌素等产品的生物转化。基因的精细表达调控是构建枯草芽孢杆菌细胞工厂的关键技术。由于启动子序列决定了其下游基因的表达强度,因此启动子元件也就成为了驱动基因表达、调控基因线路、构建人工细胞工厂的合成生物学核心元件[3]。于是,挖掘与分析枯草芽孢杆菌启动子序列结构功能及其规律,设计与构建可按需精细调控目标基因表达的启动子,具有重要研究意义和应用潜力。
启动子工程面临的挑战
常用的启动子工程策略
启动子工程主要聚焦于对天然启动子序列进行随机突变或是对功能模块的重新组合进行筛选,以获得满足需要的新启动子元件。在突变策略方面,常用方法如通过定点随机突变[4]、易错PCR[5]和非保守区随机突变[6]等方式对启动子的进行突变,以获得不同强度的(合成)启动子库;在组合启动子策略方面,常用方法如启动子的杂交[7]、转录因子结合位点(TFBS)的优化[8]等手段,此外还有启动子的串联[9]等手段。
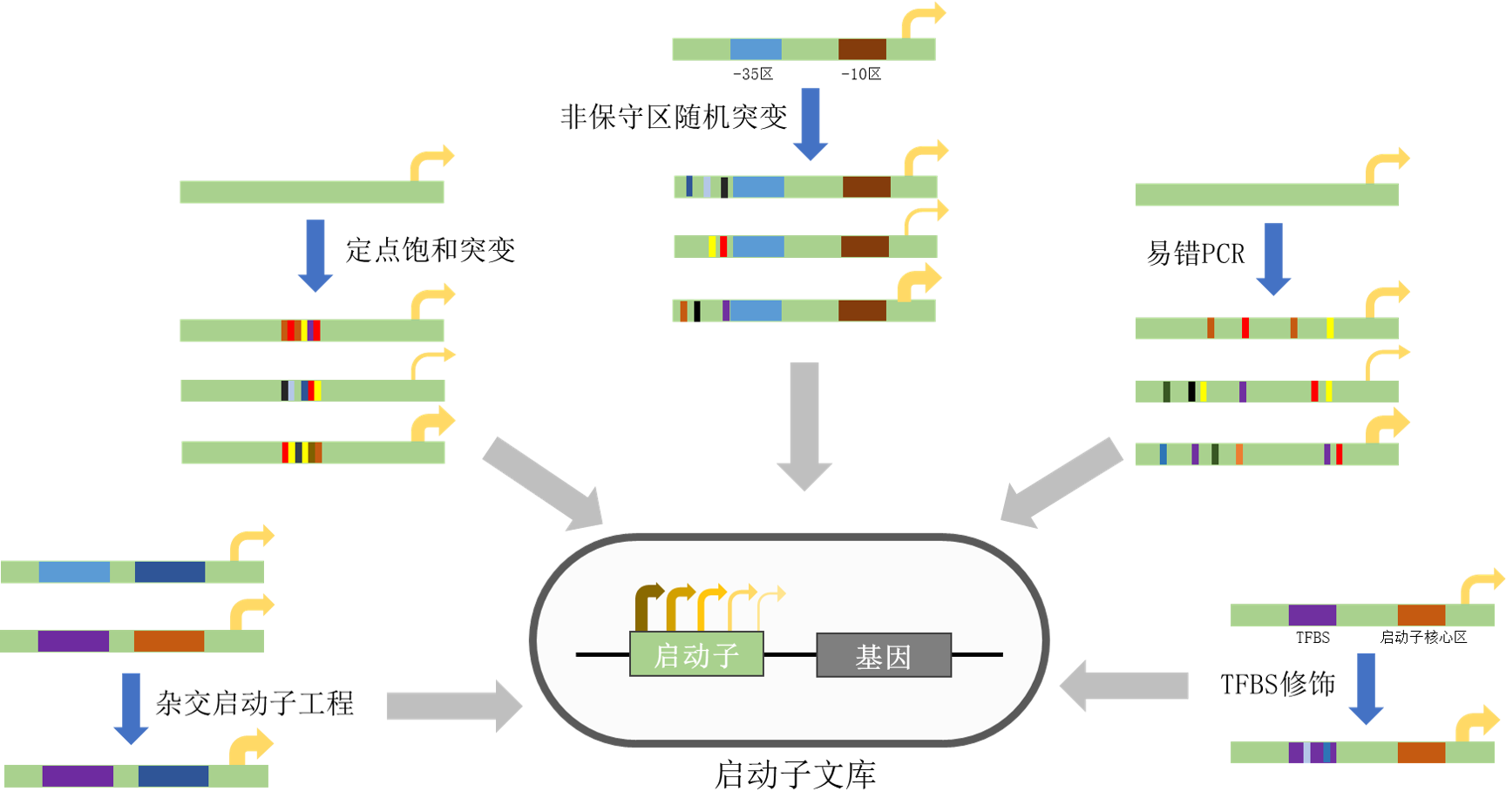
图2 启动子工程的常用工程策略
常用的启动子工程改造的局限性
随机突变策略虽然能够得到强度梯度分布的启动子库,但突变产生的潜在序列随序列长度的增加呈指数增长,筛选工作量巨大。50个碱基长的DNA序列,其潜在碱基组合为450,即存在约1030种潜在的待测序列,远超目前任何实验文库(约106~8复杂度)的筛选能力[10]。另一方面,突变产生的序列与天然序列相似度极高,易与宿主细胞产生互作关系,难以保证元件的绝缘性和遗传稳定性[11]。而启动子改造策略的虽然针对性更强,筛选工作量较小,但可供选用改造和组合的启动子通常非常有限,远不能满足研究与应用需求。
人工智能助力代谢工程与合成生物学发展
近年来,人工智能技术的迅猛发展为代谢工程与合成生物学提供了新的机遇。由于生物数据本身的高维特性以及数据中隐含模式的复杂性,深度学习算法在挖掘重要生物学特征、探求特征之间隐含的复杂关系等方面表现出了独特的优势[12]。随着各类生物大数据(如组学数据)的不断积累,人工智能技术在代谢工程与合成生物学领域展现出广阔的应用前景。
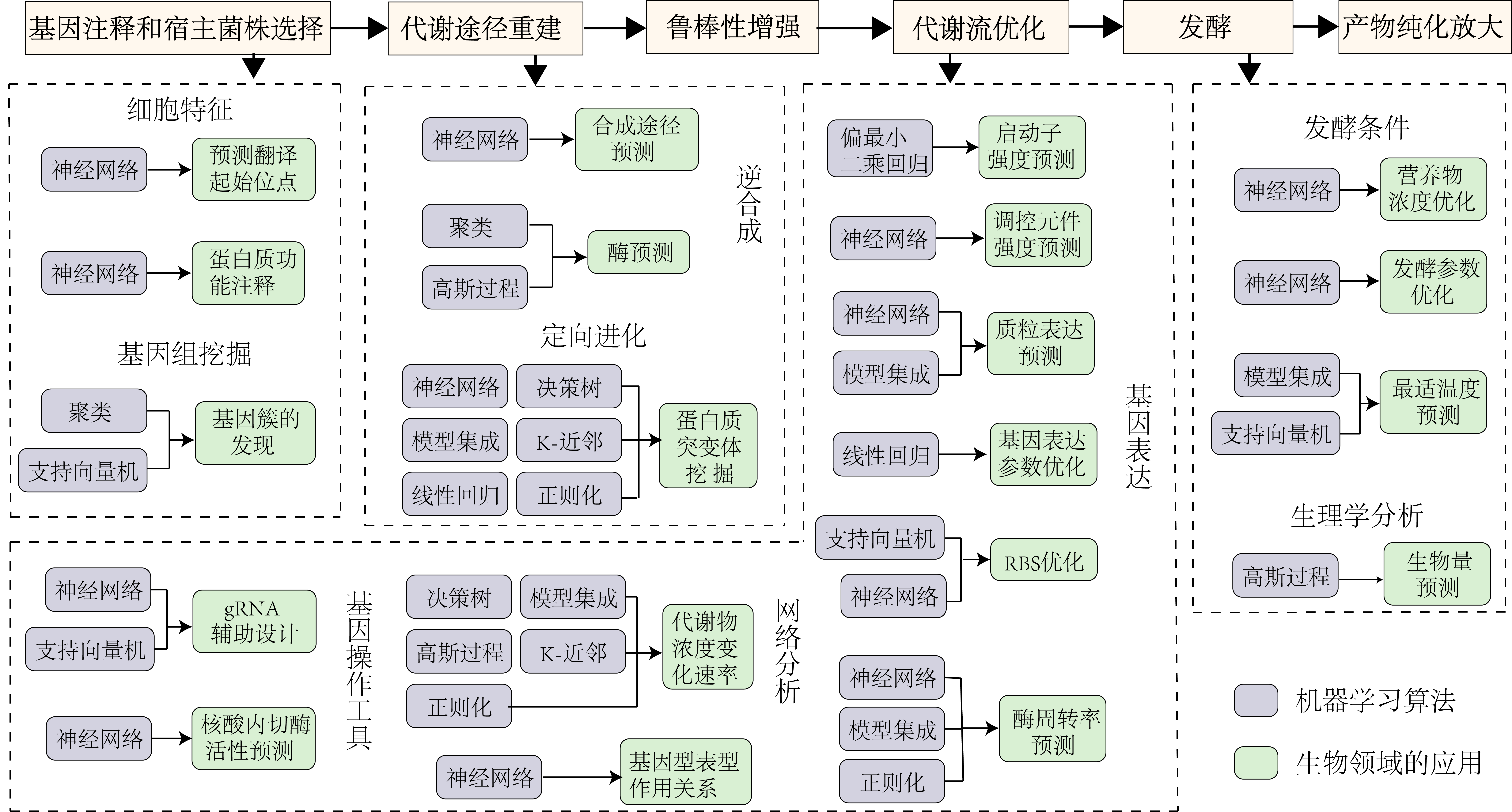
图3 基于人工智能的代谢工程与合成生物学研究
课题研究目标与意义
项目的研究目标:
(1)基于构建的预测模型对枯草芽孢杆菌启动子序列进行定量预测;
(2)基于生成模型智能设计任意强度的枯草芽孢杆菌启动子序列;
(3)解析枯草芽孢杆菌启动子序列结构与启动子强度关系的规律。
拟构建的枯草芽孢杆菌人工启动子设计分析系统,可预测与分析现有启动子序列的强度,生成给定强度的人工启动子,构建强度分布广泛的人工启动子元件库,为枯草芽孢杆菌复杂代谢网络的精准调控、酶/蛋白质的高水平表达、构建高性能菌株细胞工厂等提供重要的调控元件工具,为深入理解启动子序列与强度功能关系提供重要参考。
参考文献
[1] 于慧敏, 郑煜堃, 杜岩等. 合成生物学研究中的微生物启动子工程策略[J]. 合成生物学, 2021,
2(4):14.
[2] Ross W, Gosink K, Salomon J, et al. A third recognition element in bacterial
promoters: DNA
binding by the alpha subunit of RNA polymerase[J]. Science, 1993,
262(5138):1407-1413.
[3] Blazeck J, Alper H S. Promoter engineering: Recent advances in controlling
transcription at the
most fundamental level[J]. Biotechnology Journal, 2013, 8(1):46-58.
[4] Asakura Y, Kimura E, Usuda Y, et al. Altered metabolic flux due to deletion of
odhA causes
L-glutamate overproduction in Corynebacterium glutamicum[J]. Appl Environ Microbiol,
2007,
73(4):1308-1319.
[5] Alper H, Fischer C, Nevoigt E, et al. Tuning genetic control through promoter
engineering[J].
Proceedings of the National Academy of Sciences, 2005, 102(36): 12678-12683.
[6] Wei L, Xu N, Wang Y, et al. Promoter library-based module combination (PLMC)
technology for
optimization of threonine biosynthesis in Corynebacterium glutamicum[J]. Applied
Microbiology and
Biotechnology, 2018, 102(9).
[7] Blazeck J, Garg R, Reed B, et al. Controlling promoter strength and regulation
in Saccharomyces
cerevisiae using synthetic hybrid promoters.[J]. Biotechnology & Bioengineering,
2012,
109(11):2884-2895.
[8] Wang W, Li X, et al. An Engineered Strong Promoter for Streptomycetes[J].
Applied and
Environmental Microbiology, 2013, 79(14).
[9] Liu Z, Zheng W, Ge C, et al. High-level extracellular production of recombinant
nattokinase in
Bacillus subtilis WB800 by multiple tandem promoters[J]. BMC Microbiology, 2019, 19:
89.
[10] Urtecho G, Tripp A D, Insigne K, et al. Systematic Dissection of Sequence
Elements Controlling
σ70 Promoters Using a Genomically-Encoded Multiplexed Reporter Assay in E. coli[J].
Biochemistry,
2018:1539-1551.
[11] Rui M, Vogl T, Kniely C, et al. Synthetic Core Promoters as Universal Parts for
Fine-TuningExpression in Different Yeast Species[J]. Acs Synthetic Biology, 2017,
6(3):471-484.
[12] Meng Hailin, Ma Yingfei, Guoqin Mai, et al. Construction of precise support vector machine based models for predicting promoter strength[J]. Quantitative Biology, 2017, 5(1): 90-98.