训练数据库的构建
用于序列生成模型的天然启动子序列数据库
为了训练序列生成模型,我们使用多种启动子预测软件投票的方式对枯草芽孢杆菌4537个基因的上游序列进行预测,将集成的预测结果作为天然启动子序列数据库。该数据库包含4501条启动子序列,后续被用于VAE的训练及遗传算法中适应度函数的计算。
用于强度预测模型的序列-强度数据库
为了训练强度预测模型,我们分别构建了两个启动子序列-强度数据库,这两个数据库有着不同的强度量化指标:转录组测序获取的转录强度和利用绿色荧光蛋白表征的荧光强度。
(1)序列-转录强度数据库
我们对野生型枯草芽孢杆菌(Bacillus subtilis 168)进行转录组测序,获得了不同基因的转录强度(表示为FPKM值)。依照启动子序列与调控基因的对应关系,将基因的转录强度与预测得到的天然启动子序列数据库进行一一对应。由于不同基因的表达强度相差几个数量级,为了提升训练效果,我们将强度为0的数据舍弃并对所有数据取log2处理。去掉重复序列后,包含4128个数据的序列-转录强度数据,其中启动子的强度分布如图1所示。
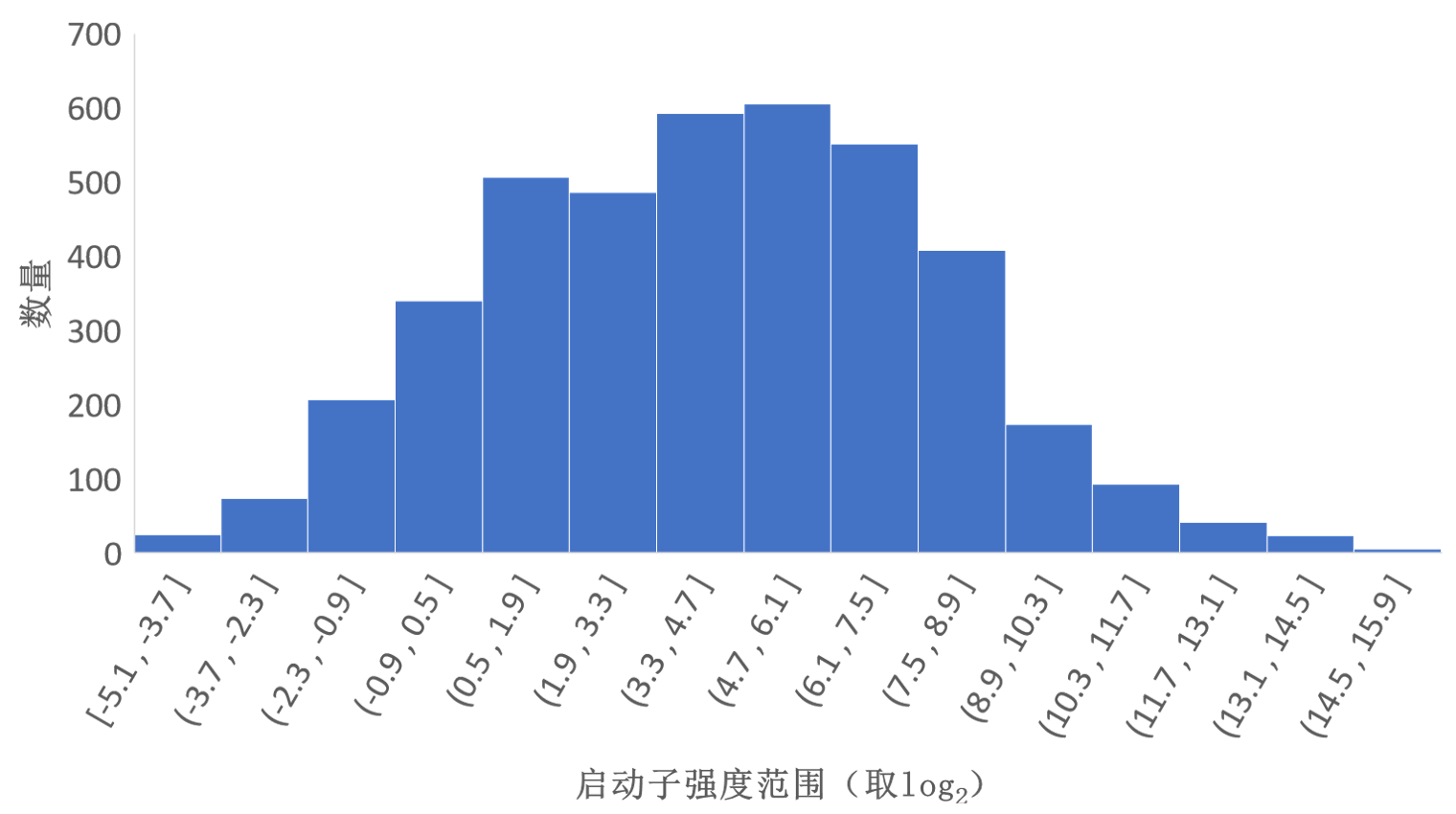
图1 启动子强度分布情况
(2)序列-荧光强度数据库
在收集天然启动子数据的同时,我们还收集了多篇在枯草芽孢杆菌中构建合成启动子文库的相关数据。这些文献均以相对荧光强度表示启动子强度,但由于不同文献表示相对强度的参考值不同,为了获得一致化的本地数据,我们从每组文献选取出中等强度的启动子,以绿色荧光蛋白作为报告基因进行实验表征,并将枯草芽孢杆菌中典型的强启动子PvegI的强度定为100。实验测得相对荧光强度如图2所示。
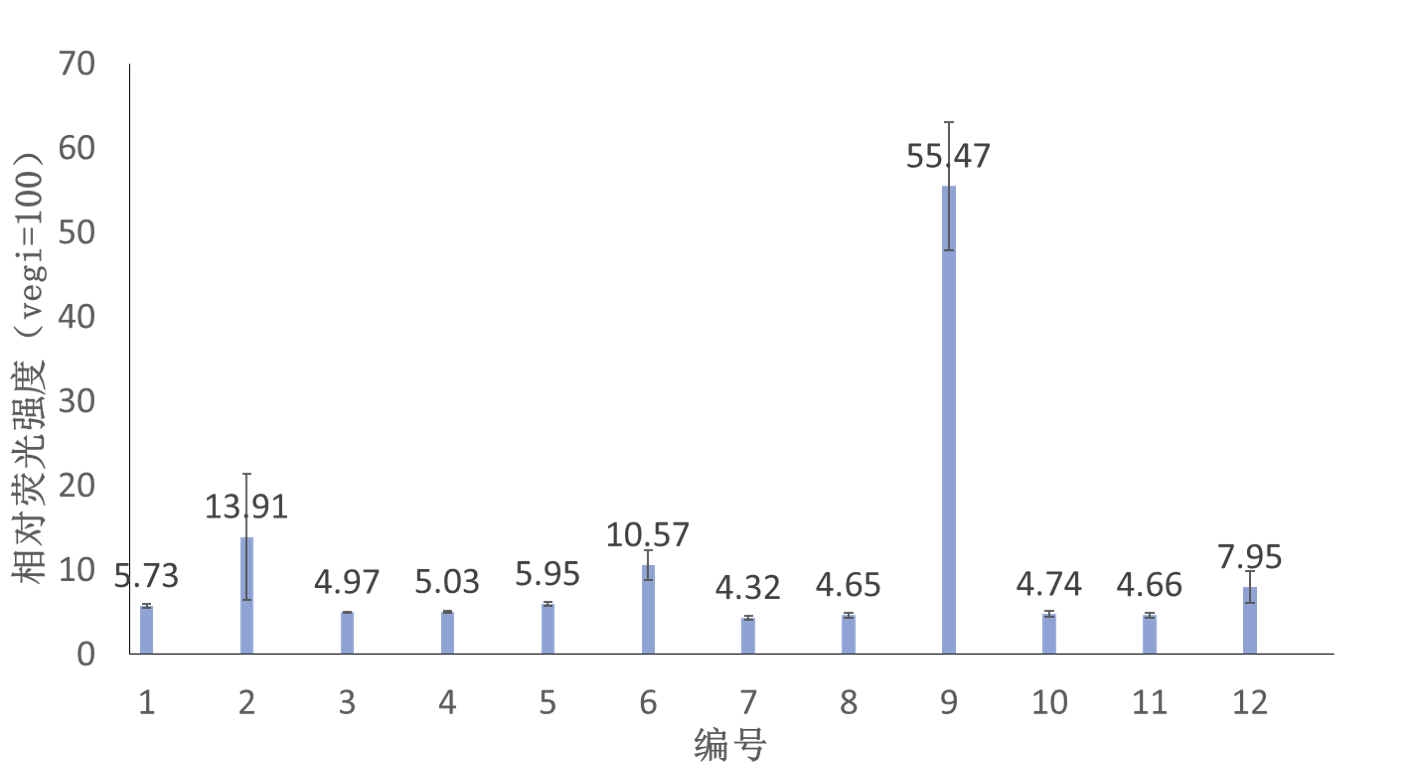
图2 文献数据的一致化处理
以实验测得的表达强度为标准将文献数据的强度进行统一,经过去除重复的启动子序列,并对其相对荧光强度处理(取log2)后,我们得到了578条经过实验验证的启动子数据集。
>> 有关数据库构建的具体过程 详见 方案页面
序列生成模型的训练与评估
变分自编码器(VAE)
如 模型 中描述,我们构建了变分自编码器(Variational auto-encoder,VAE)用于启动子序列的生成。使用天然启动子序列数据库作为训练集,将重构损失和KL散度作为损失函数,使用Adam优化器进行训练,约1000epoch后,训练损失趋于收敛(图3A)。
训练完成后,向解码器(Decoder)输入随机采样的隐含向量即可生成新的启动子。如图3B所示,图中-10和-35位置前后比其他区域稍亮一些,可见其特征更为明显。将模型输出结果取argmax后转换回DNA序列,即可获得模型生成的启动子序列。
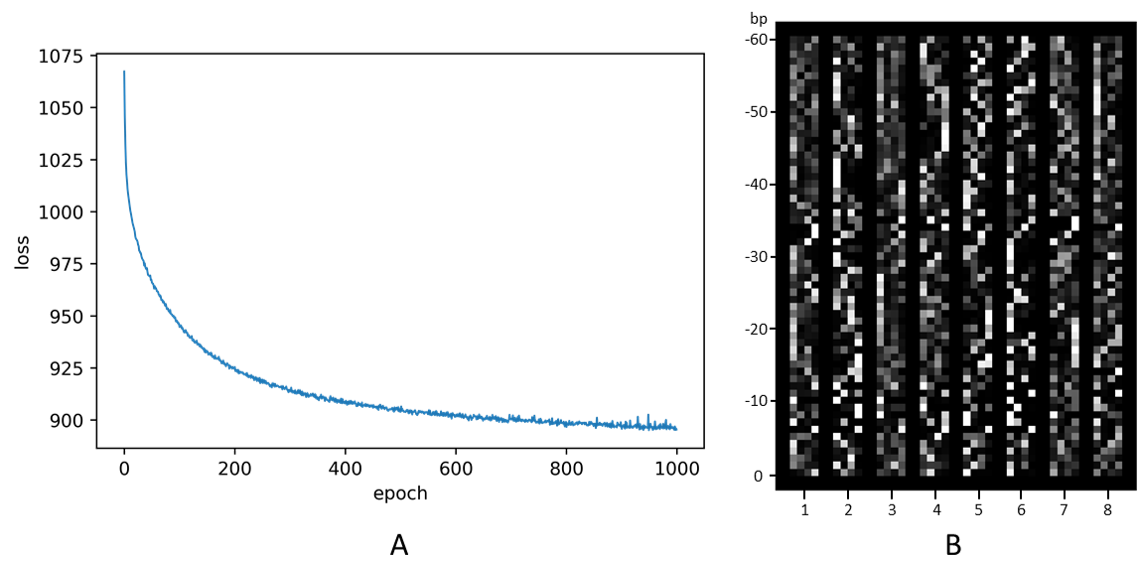
图3
VAE的训练与启动子的生成
A: VAE的训练过程; B:
VAE生成启动子的示意图,每4列分别代表一条序列中的A、C、G、T,像素点越亮表示模型对这个位置的碱基置信度越高。
遗传算法
如模型model中描述,我们还建立了用于启动子序列生成的遗传算法,在其建立过程中共实现了三个版本。为了分析和对比三个版本的启动子生成效果,我们依照表1中的参数对其进行训练,生成的结果使用WebLogo提供的在线服务绘制序列标识图(图4)。
| 版本号 | 每代个体 | 每代与目标种群整体PSSM距离 | 每代与个体聚类PSSM距离 | 强度范围 | 最终选择与整体分布距离 | 交叉概率 | 变异概率 | 代数 |
|---|---|---|---|---|---|---|---|---|
| v1.0 | 100000 | 大于1 | -- | -- | 大于20 | 0.1 | 0.01 | 50 |
| v2.0 | 100000 | 大于1 | 小于50 | -- | 大于20 | 0.1 | 0.01 | 50 |
| v3.0 | 100000 | 大于1 | 小于60 | 1~3 | 大于20 | 0.1 | 0.01 | 50 |
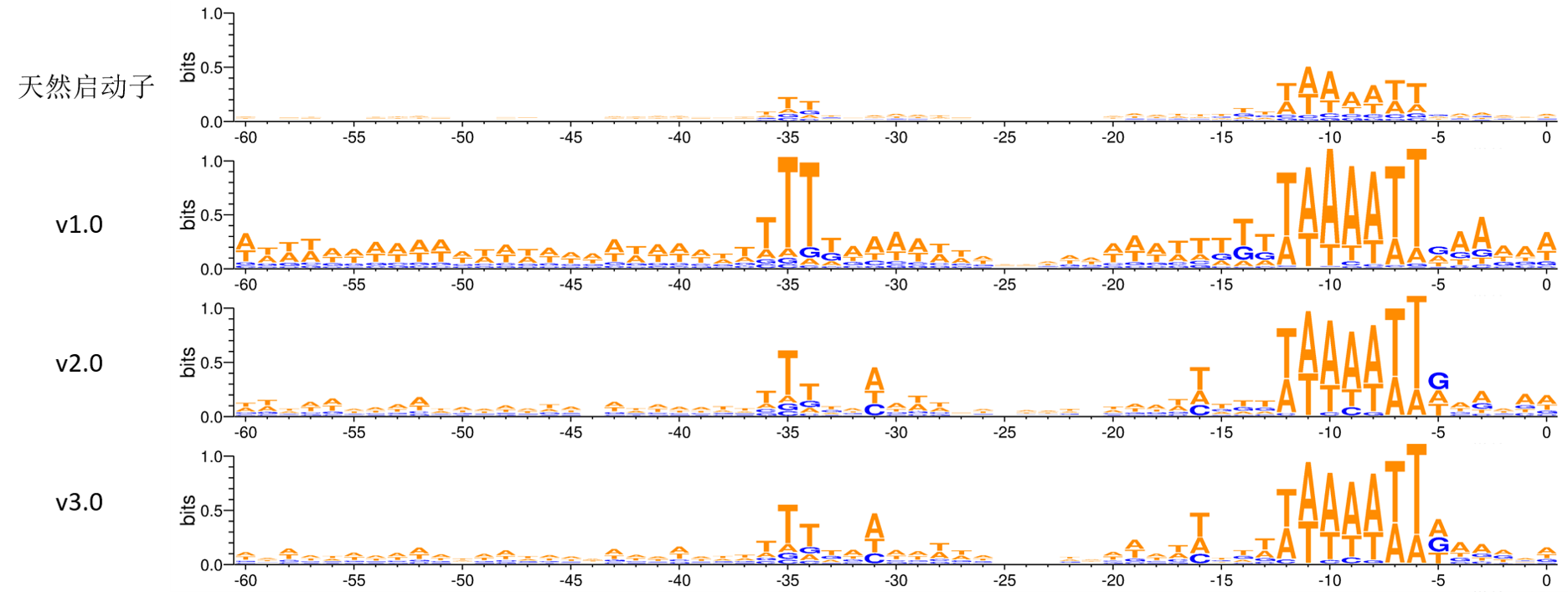
图4 遗传算法的训练结果
从图4中可以得到如下结论:
- 遗传算法能够生成与目标种群具有相似概率分布的基因序列。
- 由于fitness函数淘汰了许多与整体有轻度差异的个体,保留了大部分与整体高度相似的个体,因此提取的特征较为单一,如-10区,相比于天然启动子,许多不显著的特征被“灭绝”;
- 另一方面,由于其“进化-淘汰”的机制,一些在天然启动子中较为隐蔽的模式在生成的序列中也可以被凸显出来;
- 考虑生成序列与目标种群的个体差异后,生成序列的多样性增加,体现为图4中的标识高度降低、更接近天然启动子。
两种方式进行启动子从头设计
模型训练
强度预测模型初步构建完成后,我们分别利用启动子序列-转录强度数据库与序列-荧光强度数据库对所构建的强度预测模型进行训练。通过K-折交叉验证的结果对模型的学习速率α、迭代次数epoch、批量大小batch-size、卷积核大小与数目、嵌入层维度等参数进行调整优化。
在训练过程中,我们发现:由启动子预测识别结合转录组测序获得的启动子序列-转录强度数据库,虽然数据量较大,启动子类型更丰富,但很难利用模型训练出有价值的序列与强度的对应关系;而利用文献收集结合实验室一致化获得的启动子序列-相对强度数据库,能获得较好的训练模型,并能随着模型的改进和参数的调整,不断提高预测能力(图5)。

图5
不同数据库训练结果比较图
A:序列-转录强度数据库;B:序列-相对强度数据库
对此,我们分析有以下几点可能的原因:
- 序列-转录强度数据库的获得经过启动子预测与转录组测序两步,影响数据准确度的误差因素更多,可能导致序列与强度的相关性降低;
- 由于原核生物的多顺反子结构,一个启动子可能控制多个基因的表达,启动子强度和部分基因的转录强度关联性较低,给模型训练带来困难;
- 序列-荧光强度数据库是经实验的一致化处理,由启动子调控的GFP基因的荧光表达强度与细胞生物量的比值作为启动子的相对强度,消除了其他因素的影响,序列与强度的相关性更强。。
因此,后续我们使用启动子序列-荧光强度数据库作为模型的训练数据集。
在模型的参数调整过程中,我们在模型网络中加入Dropout层,在训练中暂时随机丢弃部分神经元以减少神经元之间复杂的共适应关系,防止过拟合现象,并加入BN(Batch Normalization)层以加快模型拟合速度,提升泛化能力。最终,确定模型的关键参数学习速率α=0.001、迭代次数epoch=300、批量大小batch-size=32,卷积核数目dim=256,嵌入层维度embedding_dim=128。
模型评估
为评估我们所构建的预测模型的定量预测能力,选用平均绝对值误差(Mean Absolute Error,MAE)以计算预测值和观测值之间绝对误差的平均值,同时选用皮尔逊系数(Pearson product-moment correlation coefficient,PCCs)以度量预测值和观测值之间的相关程度。即用MAE和PCCs作为模型预测性能的评估指标。(其中MAE的值域为[0,∞],值越小表示误差越小。PCCs的值域为[-1,1],值越大表示变量间相关性越高)
我们随机将数据集的80%数据作为训练集训练模型,将剩下的20%数据作为测试集测试模型性能,并使用K-折交叉验证(k=5)进一步验证。最终我们的预测模型在测试集上结果为MAE=0.8095,PCCs=0.6063。(效果优于2018年Wang等[1]在Nucleic Acids Research上报道的预测器性能,PCCs=0.514)

图6
强度预测模型的训练及验证结果图
A: 训练过程中的损失变化趋势; B: 训练集预测结果; C: 测试集预测结果
此外,为了进一步评估我们所构建的预测模型的性能,我们还构建了专门用于文本处理的TextCNN模型(Text Convolutional Neural Network)、经典的卷积神经网络模型Vgg16(Very Deep Convolutional Networks)、支持向量机(Support Vector Machine,SVM)和XGBoost算法(eXtreme Gradient Boosting),采用同样的训练及测试方式,作为评估模型性能的对照(图7)。结果显示,我们所构建的混合模型表现出了超过其他模型的预测准确性。
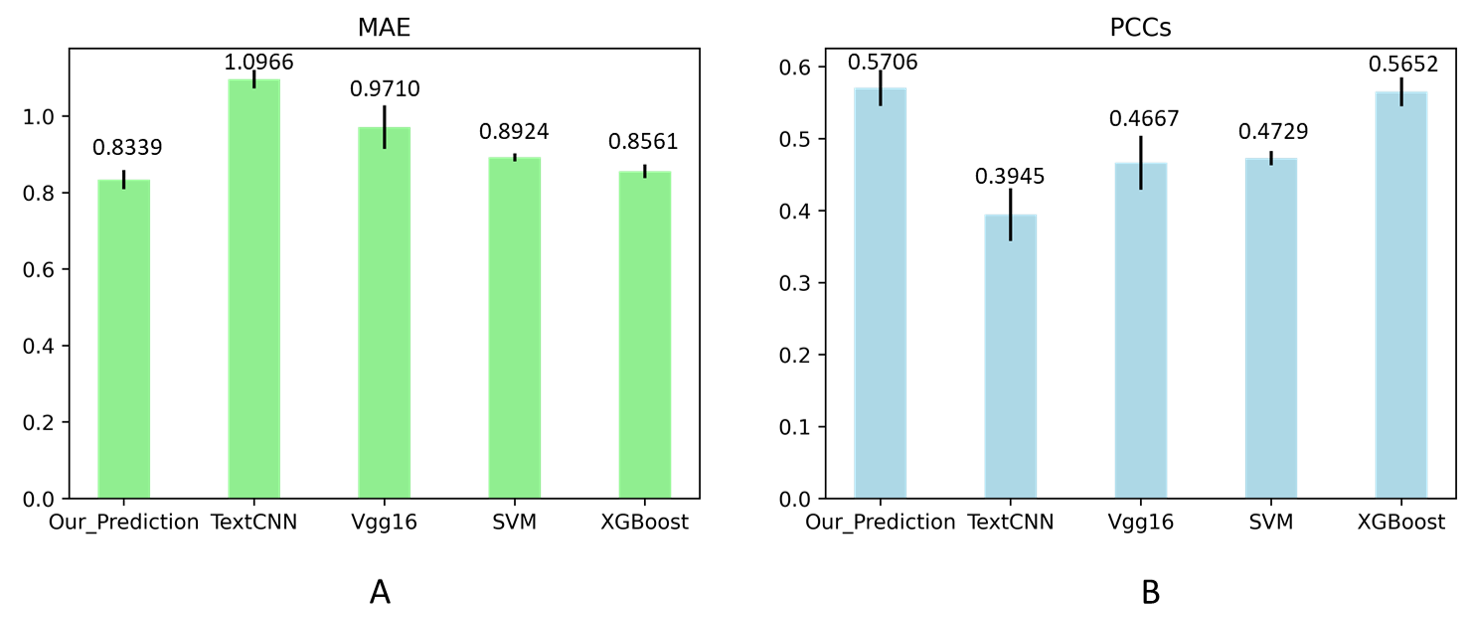
图7 多种模型预测结果图
A: 以MAE作为评价指标,值越低表示预测准确性越高; B: 以PCCs作为评价指标,值越高表示预测准确性越高
AI生成启动子的分析
AI生成启动子与天然启动子的对比
序列生成模型和强度预测模型分别构建好后,我们将其组合起来,构建完整的用于生成特定强度启动子的人工启动子设计系统。为测试该系统的效果,我们进行中等强度启动子(log2x = 1-3)的生成,并以天然启动子序列数据库作为对照,对生成的序列进行了基于基序特征的分析。生成启动子的过程如下所示。
VAE:
使用训练好的VAE生成4500个启动子序列,通过强度预测模型筛选强度为1-3的启动子。
遗传算法:
按照如下参数运行并进行筛选,最终生成了2396条序列。
| 版本号 | 每代个体 | 每代与目标种群整体PSSM距离评分 | 每代与个体聚类PSSM距离评分 | 强度范围 | 最终选择与整体分布距离评分 | 交叉概率 | 变异概率 | 代数 |
|---|---|---|---|---|---|---|---|---|
| v2.0 | 100000 | 大于1 | 小于60 | 1~3 | 大于20 | 0.1 | 0.01 | 50 |
(1)序列标识图对比
序列标识图可以通过直观的方式表示出不同位置碱基的保守性。我们使用WebLogo提供的在线服务分别生成了天然启动子、VAE生成的启动子(图中注释为VAE)、遗传算法生成的启动子(图中注释为GA)的序列标识图。
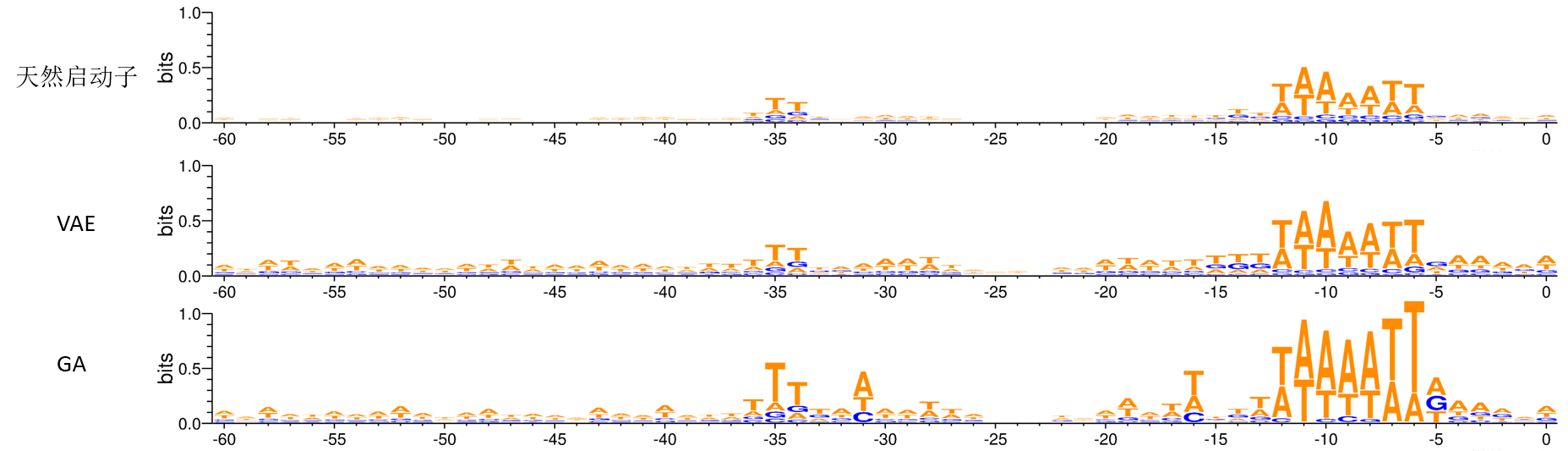
图8 合成启动子与天然启动子的序列标识图对比
从图8可以看出,在天然启动子中-10区和-35区有较为明显的特征,而这些特征也成功被我们的模型所识别到,并且相对于天然启动子其保守性更强。此外,遗传算法生成的启动子在部分位置(如-31、-16、-5bp)发现了天然启动子没有的特征,为未来进一步探究启动子序列与强度的关系提供了思路。
(2)k–mer频率对比
k-mer频率描述了序列中出现所有可能的k个核苷酸序列的频率,是生物信息分析中的一个重要的问题。下图表示了AI生成启动子与天然启动子的2、4、6、8-mer频率对比,横坐标为天然启动子的k-mer频率,纵坐标为AI生成启动子的k-mer频率,图中注释为二者之间的js散度(js散度越低表示两种分布越接近)。
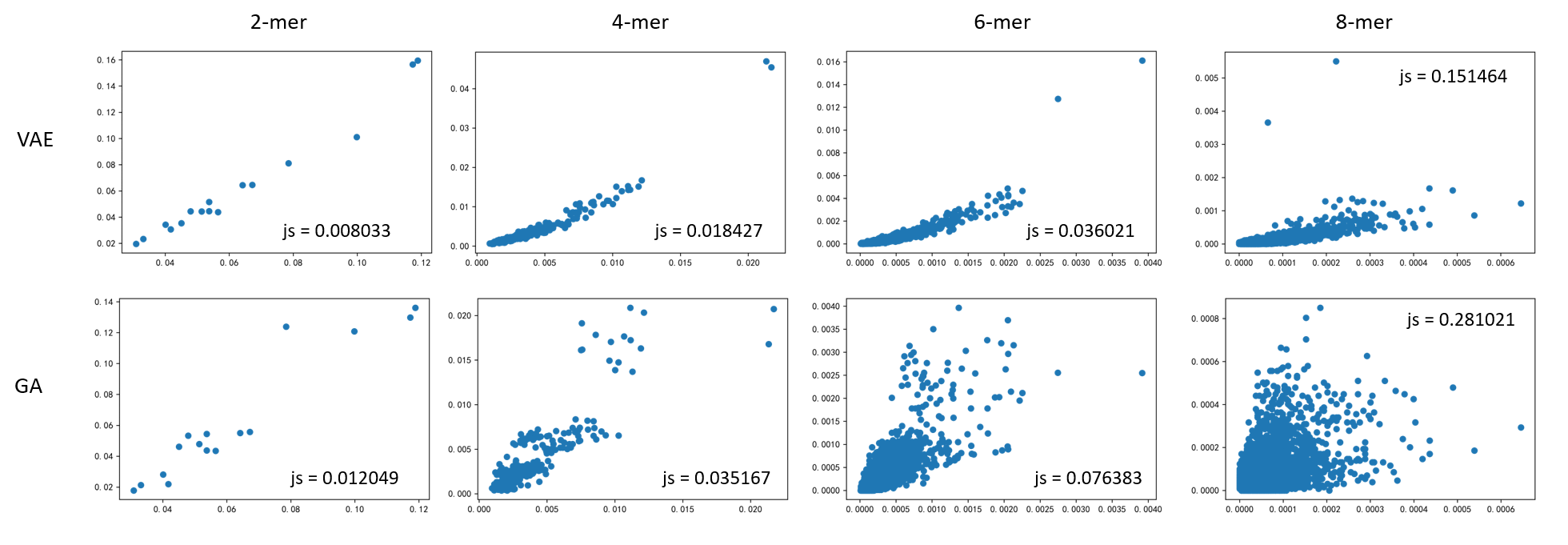
图9 AI生成启动子与天然启动子的k–mer频率对比
由于VAE在生成启动子时更能考虑到不同位置碱基之间的关系,因此其k-mer频率整体上与天然启动子更为接近。
(3)比较不同6bp基序的出现位置及频率
除了对全部k-mer频率进行统计外,我们还选取了在天然启动子中出现频率较高的几个6bp基序,分析其在序列中的出现位置,其中包括最常见的-10区(TATAAT)基序和-35区(TTGACA)基序。
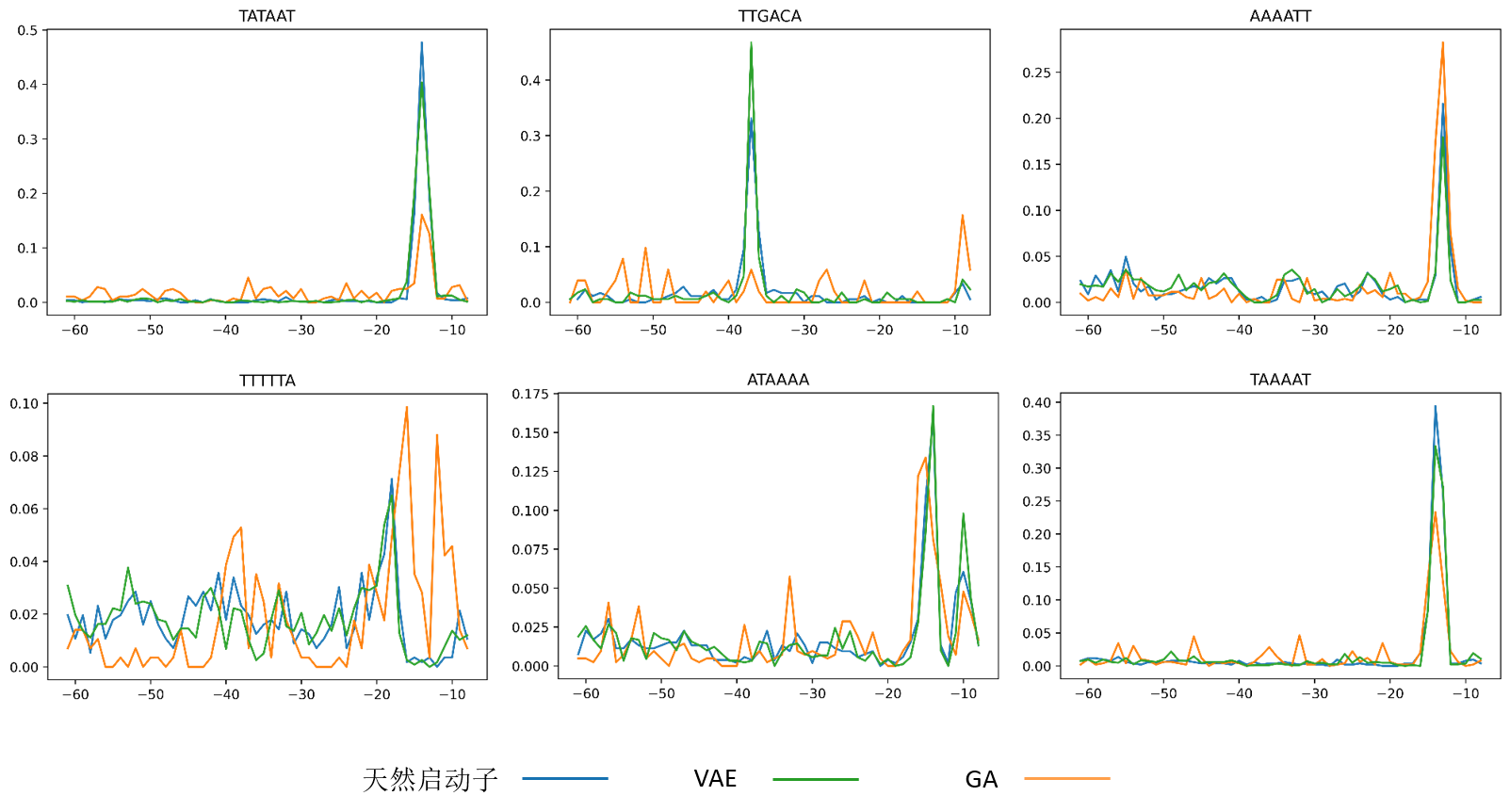
图10 不同6bp基序的位置分布
VAE对不同6bp基序拟合程度均较高,而遗传算法也能近似模拟出天然启动子的特征。另外,除了常见的-10区保守序列TATAAT外,其他一些富含A和T的序列如AAAATT、TAAAAT在-10区的出现频率也较高,这些基序可能和TATAAT同样有助于增强启动子的活性。
对模型的整体分析:
- 从统计分析的层面看,我们所生成的启动子整体上与天然启动子的特征相符,有在真正的细胞环境中发挥出启动子活性的潜力。
- 作为结构更复杂、表示能力更强的模型,VAE相比于遗传算法更能生成与天然启动子相似的序列。
- 对于遗传算法,由于其“进化-淘汰”的机制,因此更有机会将天然启动子中原本较为隐蔽的特征凸显出来,也具有独特的应用潜力。
对启动子序列与强度关系的探究
为了探究不同强度启动子在序列特征上的差异,我们使用与真实启动子较为接近的4500个VAE生成序列,使用强度预测模型对强度其进行预测,并选取强度排前500(强度范围在3.2-6.7)的启动子作为强启动子、强度排后500(强度范围在0.2-1.8)的启动子作为弱启动子进行分析。
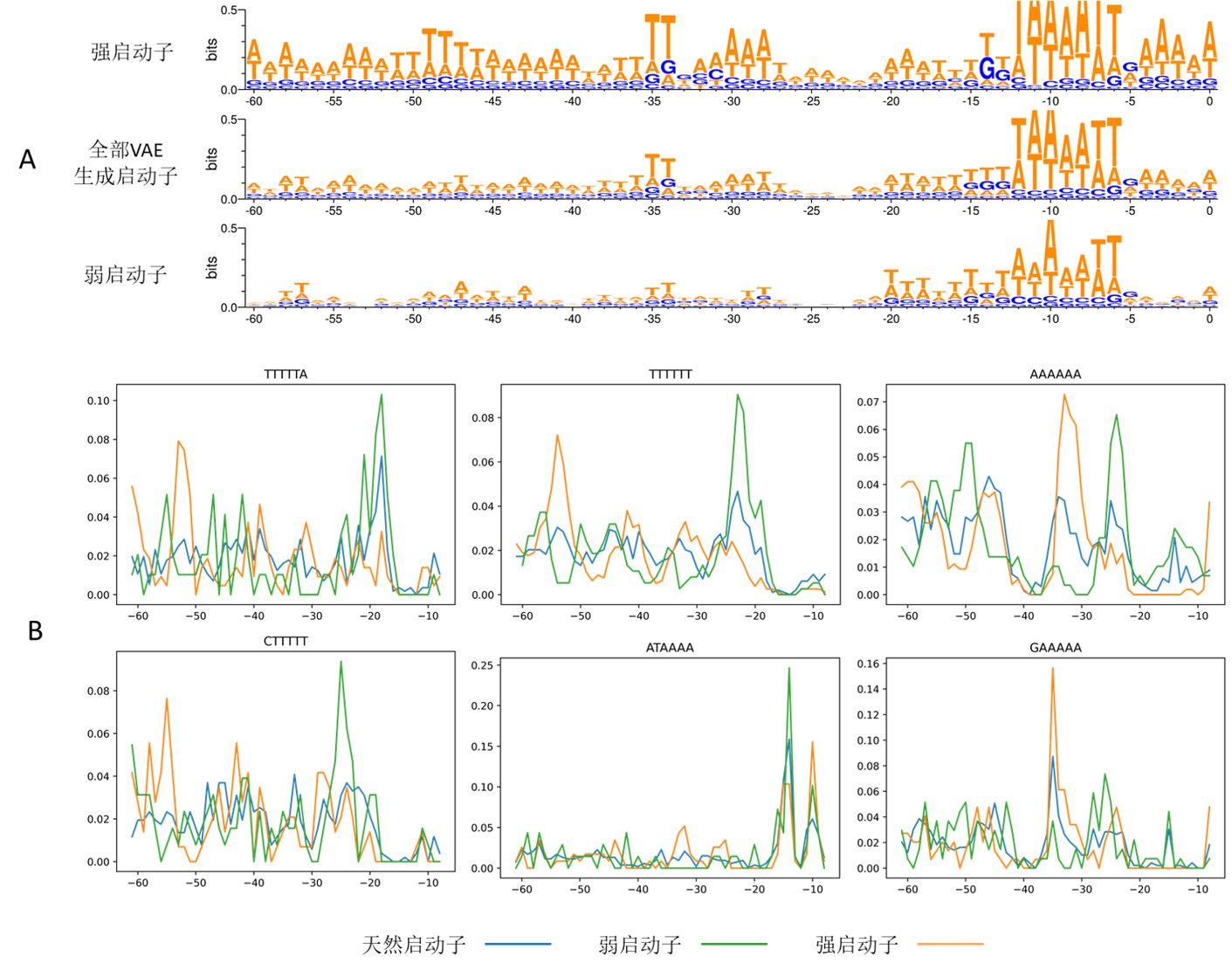
图11 对强启动子和弱启动子的分析
A:序列标示图对比;B:不同6bp基序出现频率的对比
从图11可以看出,强启动子和弱启动子有着明显的差异。强启动子的序列保守性更强,而弱启动子的序列保守性低于整体水平。并且在不同6bp基序的位置分布上,强弱启动子也有着明显的区别。
由图11A,强启动子在-10区的上游出现了一个TG二核苷酸基序,而-35区的上游出现An序列(-45至-40)和富含AT的序列(-60至-46),这均和已有的文献报道报道相互印证[2]。为排除强度预测模型训练过程中的随机性的影响,我们进行了多次重复,在结果中均发现了上述特征。
而对于图中出现的其他一些特征,如强启动子在-35位置前后出现的富含A的序列和在-55位置前后出现的富含T的序列、弱启动子在-57和-58位置的TT序列是否与其强度相关,需要待后续通过实验的方式进一步确认。
讨论
数据相关性分析
在实验的过程中,我们发现通过文献调研和实验表征得到数据库尽管数据量较小,但相对于大量的使用转录组测序和启动子预测所获得的数据而言,其训练效果明显更好。我们曾尝试使用包括Vgg16在内的更复杂、学习能力更强的模型进行训练,但没有得到更好的效果,这说明限制模型效果的因素很可能在于数据本身的相关性。
由于基因的表达强度受多种因素影响,启动子虽然在其中起到了较为重要的作用,但使用转录组测序的数据会受其他因素影响,进而使序列和强度之间的相关性降低。使用荧光蛋白虽然表征的是蛋白表达强度而非转录强度,但由于表达载体在构建过程中排除了其他干扰项,因此启动子序列和表达强度之间的相关性更强。未来我们应着眼于寻找更多具有更高相关性的数据进行训练,以达到更好的效果。
>> 这与我们咨询相关专家得到的反馈相符 详见 合作页面
AI生成启动子的分析
从序列保守性、k-mer频率、典型基序的出现位置等角度来看,AI生成启动子整体上与天然启动子有着相似的特征,并且在强启动子中也显示出了与文献报道一致的特征。
但在分析的过程中,AI生成启动子也显示出一些与天然启动子有所不同的特点,如不同位置碱基的保守性更强、特定基序在某些位置的出现频率更高,这是否与启动子的强弱相关联,需要待后续通过实验的方式进一步确认。
合成启动子活性的验证分析
我们原计划在模型构建完成后,通过选取生成的启动子重新构建GFP表达质粒的方式进行实验表征,以进一步验证模型预测准确度、探索启动子强弱规律,并指导我们对模型进行下一步的完善。但由于2021年1月天津市突发新冠疫情,所需的实验材料因学校封闭管理而无法送达,因此实验计划受到影响。待实验室恢复正常运行后,我们将完成进一步的实验分析。
参考文献
[1] Wang Y, Wang H, Wei L, et al. Synthetic promoter design
in Escherichia coli based on a deep generative network[J]. Nucleic Acids Research,
2020, 48(12): 6403-6412.
[2] Voskuil M I, Chambliss G H. The −16 region of Bacillus subtilis and other
gram-positive bacterial promoters[J]. Nucleic Acids Research, 1998. 26(15):
3584-3590.